检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
DNA复制是生命体最基础的生物学过程,控制着遗传信息的精确传递。复制源激活形成的一对“Y”形姐妹复制叉(sister replication forks),以相反方向进行复制,直至与另一个相向行进的复制叉相遇,发生复制终止(1)。早期,领域内对DNA复制的理解主要建立在一些简单模式生物的研究上,核心问题在于理解复制起始、延伸和终止过程以及复制进程的细胞周期调控,并成功利用芽殖酵母的纯化蛋白在体外重建了真核生物的DNA复制起始过程,实现了从复制源识别到复制叉形成的整个生化过程(2)。
尽管在模式生物中的研究取得了瞩目进展,我们对高等哺乳动物DNA复制的理解仍然只是冰山一角。DNA复制发生在染色质上,因而会受到染色质环境及其它DNA代谢过程的调控。酵母的DNA含量仅为人细胞的1/500,且仅具有相对简单、初步的染色质结构(3),而高等哺乳动物的基因组巨大,其染色质高度折叠,在细胞核内部形成庞大的“染色质疆域”,其中每条染色体都具有十分复杂的层级结构(4)。在这样的情况下,执行DNA复制的分子机器并非孤立存在,而是可能在空间上相互靠近,形成“复制工厂(replication factory)”,有规律地执行复制过程(5)。因此,哺乳动物的DNA复制既会受到染色质环境的调控和约束,又需要有组织地运行,不断协调与其它DNA代谢事件的关系,在保证DNA和表观遗传信息稳定遗传的同时维护基因组的稳定性(5-7)。随着研究技术的发展和研究数据的积累,在未来,系统性、整体性地研究人类复杂染色质环境下的DNA复制将是一个必然趋势。哺乳动物复制系统的复杂性给研究带来了巨大挑战,也致使领域内对人类自身DNA复制的独特机制了解尚不深入。此外,高通量、高分辨率研究方法也缺乏,导致目前这一领域的发展仍然处于初级阶段。
2024年3月15日,金沙威尼斯欢乐娱人城和北大-清华生命科学联合中心胡家志课题组在Science发表了题为Fork coupling directs DNA replication elongation and termination的研究论文,该研究建立了首个检测DNA复制叉附近染色质相互作用的方法,并发现在人类和小鼠细胞的DNA复制过程中,复制叉并非传统模型中认为的独立反方向前进,而是彼此之间能够以偶联的形式持续相互作用,协同完成整个复制延伸过程,并指导复制终止(图3)。

课题组首先建立了一种名为Repli-HiC的高通量测序方法,该方法利用核苷类似物BrdU瞬时标记复制过程中的细胞,并结合ChIA-PET和原位Hi-C的技术原理检测复制过程中新生DNA区域相关的染色质相互作用。通过Repli-HiC得到的染色质互作矩阵,研究者在人类K562细胞和小鼠胚胎干细胞中观察到垂直于互作矩阵对角线的染色质互作信号,其形状类似于喷泉结构,故将其命名为染色质喷泉,并利用新开发的生物信息学算法Fun,在基因组范围内鉴定出了数千个喷泉位点(图1)。
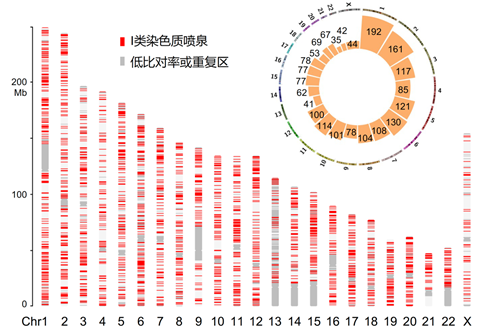
图1. Fun算法鉴定到的染色质喷泉位点在基因组上的分布情况。
结合课题组先前开发的基于核苷类似物插入检测DNA复制起始事件的测序方法NAIL-seq(7)以及用于鉴定DNA复制事件的冈崎片段测序OK-seq(8),该研究进一步发现染色质喷泉的形成可能和DNA复制过程紧密联系,并根据它们的结构特征和形成模式将其划分为两大基本类型:第一类和第二类染色质喷泉。其中,第一类喷泉结构的延伸长度较短(中位数为160kb)、宽度较窄,结构的初始位置和复制起始区域相对应,而延伸的末端与复制终止区共定位。该结构暗示着从同一个起始源发生的一对姐妹复制叉从复制的起始位置开始,便以偶联的形式持续地相互作用在一起,按照大致相等的速度进行双向复制,直至复制终止区域(图2 A和2B)。同时,这类喷泉结构也暗示着姐妹复制叉双生子(replication twinsome)结构可能是复制工厂的基础单元,为复制工厂理论提供了重要的分子证据。第二类喷泉的延伸长度较长(中位数为1Mb左右)、宽度较宽,且喷泉结构的中心坐落于复制终止区域发生的位置,两侧末端则存在DNA复制起始事件。该结构表明来自于两个不同复制起始区域的复制叉,也在染色质相互作用下有机会成对偶联,由两侧向中心区域相向行进,直至到达复制终止区域(图2 C和2D)。这表明DNA复制终止是一个预决定的事件,而非一个随机发生的复制叉碰撞事件(图3)。
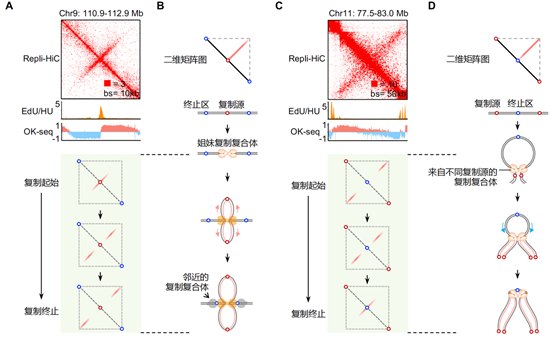
图2 DNA复制叉偶联。
A-D.偶联的姐妹复制叉(A和B)和相向行进复制叉(C和D)分别形成第一类和第二类染色质喷泉结构。在A和C中,EdU/HU鉴定早期复制起始;OK-seq鉴定复制起始(由负到正)和终止(由正到负)。浅绿色背景中展示了Repli-HiC分析复制起始、延伸和终止阶段的二维矩阵示意图。
综上,该研究是领域内首次在高等哺乳动物细胞中系统解析DNA复制叉在染色质环境下成对偶联延伸的空间组织模式,不仅支持复制工厂的基本模型,提出了DNA复制终止的预决定模型,颠覆了传统教科书中的模型(图3),更为系统性研究DNA复制、基因组稳定性和相关生理疾病的发生提供了全新的视角和理论基础,同时引申出一系列重要的科学问题:复制叉偶联的结构基础、分子机制及背后的生理学意义是什么,复制叉的偶联过程如何受到染色质环境及其它DNA代谢事件的调控等。总之,该研究为DNA复制领域铺平了道路。

图3. 复制的经典模型和本工作提出的复制叉的偶联模型。
传统模型认为相互分离的DNA复制叉执行复制延伸和终止。本工作发现DNA复制叉在整个复制过程中并不是孤立的,而以互相偶联的形成指导DNA合成。
该工作由金沙威尼斯欢乐娱人城和北大-清华生命科学联合中心胡家志研究员、李晴教授、孔道春教授、高宁教授和季雄研究员组合作完成,胡家志研究员为该论文的通讯作者。金沙威尼斯欢乐娱人城博士后刘阳博士、2021级博士研究生张丁峥嵘及刘栩豪为该论文的共同第一作者。甘婷婷博士、艾晨博士、吴锦淳博士、梁昊昕、陈莫晗、郭岳峰、卢如森、蒋永鹏博士等在该工作中亦有重要贡献。该研究还得到了金沙威尼斯欢乐娱人城吴虹教授、中科院生物物理所朱冰研究员和浙江大学生命科学研究院阮一骏教授的帮助和指导。该工作得到了国家自然科学基金、科技部及农业部的支持。
原文链接:https://www.science.org/doi/10.1126/science.adj7606
胡家志实验室网址:https://hulab.pku.edu.cn/
参考文献:
1 O'Donnell, M., Langston, L. & Stillman, B. Principles and concepts of DNA replication in bacteria, archaea, and eukarya. Cold Spring Harb Perspect Biol 5, doi:10.1101/cshperspect.a010108 (2013).
2 Gasser, S. Once and Only Once. Cell 177, 495-498, doi:10.1016/j.cell.2019.03.034 (2019).
3 Shao, Y. et al. Creating a functional single-chromosome yeast. Nature 560, 331-335, doi:10.1038/s41586-018-0382-x (2018).
4 Finn, E. H. & Misteli, T. Molecular basis and biological function of variability in spatial genome organization. Science 365, doi:10.1126/science.aaw9498 (2019).
5 Vouzas, A. E. & Gilbert, D. M. Mammalian DNA Replication Timing. Cold Spring Harb Perspect Biol 13, doi:10.1101/cshperspect.a040162 (2021).
6 Wu, J. et al. Cohesin maintains replication timing to suppress DNA damage on cancer genes. Nat Genet 55, 1347-1358, doi:10.1038/s41588-023-01458-z (2023).
7 Liu, Y. et al. Transcription shapes DNA replication initiation to preserve genome integrity. Genome Biol 22, 176, doi:10.1186/s13059-021-02390-3 (2021).
8 Petryk, N. et al. Replication landscape of the human genome. Nat Commun 7, 10208, doi:10.1038/ncomms10208 (2016).