检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
2021年9月21日,金沙威尼斯欢乐娱人城和金沙威尼斯欢乐娱人城生态研究中心姚蒙和李晟课题组在Current Biology上以研究长文(Article)发表题为“Prey partitioning and livestock consumption in the world’s richest large carnivore assemblage”的论文。该论文围绕全球生物多样性热点区“中国西南山地”,通过对3个山系生态系统中多种食肉动物的1000余份粪便样品的DNA metabarcoding(宏条形码)分析,深入解析同域共存的食肉动物的食性特征,量化构建捕食者-猎物的食物网结构。结果显示,不同食肉动物物种间存在不同程度的食性生态位分化,这可能是实现群落内物种稳定共存的重要机制。在藏族牧区,家畜在大型食肉动物的食物中占比较高,提示在人类活动占主导作用的景观中,文化包容性可能对大型食肉动物种群延续起到了重要作用。

中国西南山地拥有全球多样性最高的食肉动物群落(照片为研究团队红外相机野外实地拍摄)
陆地生态系统中食肉动物物种如何在群落内共存,以及捕食者与猎物之间如何构建复杂的食物网结构,是动物生态学研究的核心问题之一。地处青藏高原东缘的中国西南山地是全球大型食肉动物多样性最高的区域,为研究复杂生态系统中食肉动物之间以及捕食者-猎物之间的种间关系和共存机制提供了理想的研究场所。
本研究以中国西南山地的沙鲁里山、邛崃山和岷山山脉为研究区域,在野外系统收集1,706份食肉动物粪便样品,成功鉴定出食肉动物粪便1,185份,确定来源于17种食肉目哺乳动物;从其中1,097份样品通过基于粪便DNA metabarcoding技术获得7.44亿条序列数据,检测到分属12目的95种脊椎动物食物种类。利用分子食性分析结果,针对中国西南山地食肉动物群落进行定量食性组成、食性生态位关系和食物网结构的分析。
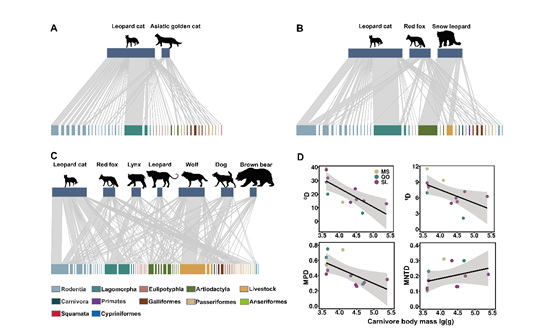
西南山地3个山系的生态系统内食肉动物-猎物的量化食物网结构
A. 岷山;B. 邛崃山;C. 沙鲁里山;D.食物多样性与食肉物种体重的关系
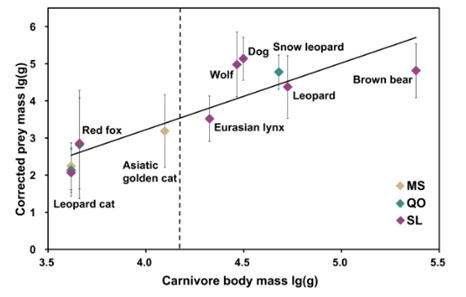
食肉动物的加权食物物种体重与其体重呈线性正相关
结果显示,不同食肉动物物种的食物物种多样性和系统发育多样性呈现随其自身体重增加而减小的趋势特征;食肉动物与其猎物的体重之间存在正相关性,体型相近的食肉动物之间具有更高程度的食性重叠。在大型食肉动物最为丰富的沙鲁里山系,有蹄类家畜在野生大型食肉动物和狗的食性中均占重要比例,同时狗也捕食一定量的野生猎物;在这一区域,我国西部地区传统文化的积极作用,使得当地居民对于食肉动物捕食家畜具有较高的耐受力与包容性,一定程度上缓解了人兽冲突的激烈程度。

沙鲁里山大型食肉动物对家畜具有强烈取食偏好
本研究基于分子食性技术和大样本量的分析,在全球首次量化构建了食肉动物-猎物复杂食物网结构(食肉动物物种数>3),阐明了复杂森林生态系统中食肉动物之间的食性生态位关系及其与猎物之间食物网结构。研究为中国西南山地多种食肉动物的食性特征及食性生态位关系提供了关键信息,发现食性分化与家畜来源的食物补充是西南山地食肉动物群落物种共存的重要机制。本研究开发并检验了陆地生态系统中捕食者-猎物复杂食物网构建的技术与方法,研究结果对理解食肉动物共存机制具有重要的科学意义,丰富并完善了兽类群落中食肉动物物种共存机制的理论基础,同时对野生食肉动物多样性的保护、缓解人兽冲突并促进人类与野生食肉动物共存具有重要的参考价值。

论文第一作者邵昕宁(左)和沙鲁里山地区喇嘛(右)在野外采集样品

姚蒙(左)、李晟(右)在沙鲁里山采集兽类粪便样品

野外工作得到当地保护区工作人员的大力协助
姚蒙、李晟为本论文的共同通讯作者。金沙威尼斯欢乐娱人城博士生邵昕宁为论文的第一作者。该研究得到国家自然科学基金、青藏高原二次科考项目、生态环境部及香港海洋公园保育基金的支持。野外工作得到相关自然保护区与四川省林业和草原局的大力协助。

姚蒙:
金沙威尼斯欢乐娱人城、金沙威尼斯欢乐娱人城生态研究中心赵进东课题组副研究员
实验室研究领域:
1. 利用分子宏条形码和环境DNA技术进行生物多样性研究
2. 野生濒危动物种群遗传学
李晟:
金沙威尼斯欢乐娱人城、金沙威尼斯欢乐娱人城生态研究中心研究员
实验室研究领域:
1. 大型兽类生态与保护
2. 生物多样性监测与自然保护地管理
原文链接:https://www.cell.com/current-biology/fulltext/S0960-9822(21)01203-3#relatedArticles