检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
2021年5月17日,金沙威尼斯欢乐娱人城陆剑课题组在《WIREs RNA》上发表了题为“Evolutionary driving forces of A-to-I editing in metazoans”的综述论文。
由ADAR(adenosine deaminase acting on RNA)蛋白介导的腺嘌呤到次黄嘌呤(A-to-I)的RNA编辑是后生动物中广泛存在的转录后修饰。由于I会被识别为G,因此A-to-I RNA编辑在不改变基因组序列的情况下,时空特异性地增加了转录组和蛋白组的多样性。陆剑课题组之前的工作已经报道了在果蝇中存在大量改变氨基酸的非同义RNA编辑位点(Nonsyn),这些编辑位点呈现出适应性信号,受到正向自然选择(Duan et al., 2017, PLoS Genetics),并且这些成簇分布的、具有适应性的非同义RNA编辑事件倾向于连锁在相同mRNA分子上,同时被编辑(Duan et al., 2018, Molecular Biology and Evolution)。随后,课题组又报道了果蝇和蜜蜂里A-to-I编辑的趋同适应性演化:虽然编辑位点的位置不保守,但很多发生在相同基因上,并在两个物种中都呈现出适应性信号(Duan et al., 2021, iScience)。
在该综述中,作者总结该领域相关进展,探讨了ADAR基因的演化、A-to-I RNA编辑位点在后生动物中的分布和演化驱动力、非同义编辑位点的生物学意义,以及RNA编辑的趋同演化现象。
基因复制事件是新基因产生的重要来源。ADAR基因、tRNA编辑酶ADAT基因,以及不具编辑活性只在睾丸中表达的ADAD基因就是由一系列基因复制事件产生的(图一)。ADAT基因最古老,在后生动物与原生动物中都存在。而ADAR与ADAD的同源性更高。由于ADAR在后生动物中都存在,ADAD只在脊椎动物中存在,因此最可能的演化历程是,在后生动物与原生动物分歧之后,ADAR由ADAT复制产生,并在脊椎动物中由ADAR演化出了ADAD。
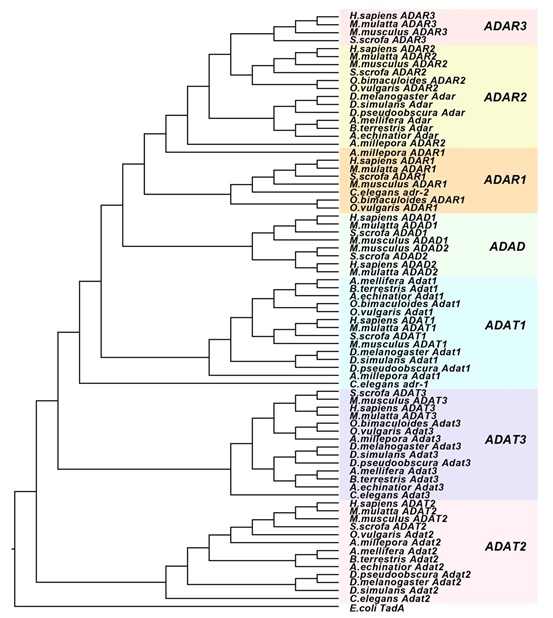
图一,ADAR、ADAD与ADAT基因的演化关系。
同时,在演化过程中ADAR自身也经历复制与丢失事件。哺乳类有3个ADAR,其中ADAR1主要负责重复序列(如灵长类Alu)的编辑,ADAR2主要负责mRNA编码区的编辑,ADAR3没有编辑活性。ADAR1与ADAR2来源于基因复制事件,而ADAR3是由ADAR2进一步复制产生的。昆虫中则是丢失了ADAR1,仅存在一个Adar,与哺乳类ADAR2同源(图一)。
在编辑位点有详尽研究的16个代表性后生动物物种中,编辑位点数量以及分布规律很不一样(图二)。例如,哺乳动物中编辑位点大部分位于重复序列,而果蝇和头足类动物中,编码区的位点占了很大比例。因此接下来作者探讨了在后生动物中RNA编辑位点的演化驱动力,认为以下这些因素可能造成不同物种中编辑位点的差异。
首先是ADAR基因的种类。哺乳动物中有介导重复序列编辑的ADAR1,而果蝇中丢失ADAR1,仅有负责CDS编辑的ADAR2同源基因。ADAR基因的差别能够部分解释RNA编辑位点在这些进化枝上的分布差异。
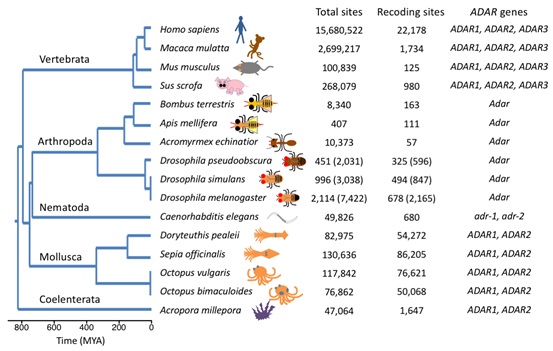
图二,16个代表性后生动物的演化关系、编辑位点数量分布,以及ADAR基因种类。
其次,ADAR基因表达模式不同。哺乳类ADAR1与ADAR2在多种组织中表达,可能产生非特异性的过度编辑,这些编辑位点大多是中性或轻微有害的。而果蝇的Adar主要在神经组织中表达,编辑位点也是富集在神经相关基因上。
第三,由于RNA编辑依赖于RNA二级结构和位点前后的序列特征,因此凡是影响这些顺式作用元件的DNA突变都会驱动编辑位点的演变。
第四,不同物种的基因组组成也是决定编辑位点分布的一大因素。哺乳动物基因组中重复序列比例很高(人类55%),而编辑位点进一步富集于重复区域(人类98%的编辑位点在重复区域)。相比之下,黑腹果蝇基因组中重复序列比例较低(22%),而编辑位点则更加规避重复区域(16%的位点在重复区域)。
第五,不同物种的有效群体Ne大小不同,使得自然选择力度不同。黑腹果蝇的有效群体远大于人类,因此果蝇中的正选择与负选择都更有效,有利的RNA编辑位点更倾向于保留,有害的编辑位点更快被去除。
接着,作者讨论了改变氨基酸的非同义编辑位点(Nonsyn)的生物学意义。在众多的非同义编辑位点中,除了极个别有详尽功能研究的位点之外,大部分位点的功能是未知的。现有的两种互补的理论认为,非同义编辑位点一方面可以增加转录组和蛋白组的多样性,另一方面可以弥补有害的DNA突变(图三)。
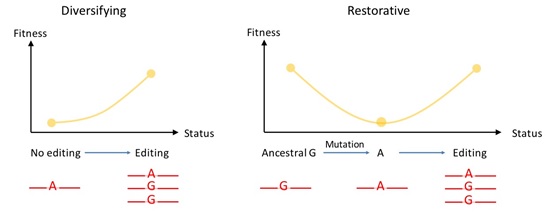
图三,两种互补的理论解释非同义编辑位点的生物学意义。
在果蝇中,非同义编辑位点的出现频率和编辑水平都要高于同义编辑位点,说明非同义编辑位点是具有演化适应性的。并且,非同义编辑倾向于发生在演化上保守的区域,这进一步支持RNA编辑的适应性假说,保守区域的DNA突变受到限制,而RNA编辑在转录后水平时空特异性地增加了蛋白多样性。在头足类动物中,数以万计的非同义编辑位点被鉴定到,并且这些位点附近的基因组突变很少。由于RNA编辑依赖于二级结构,而突变可能破坏二级结构,因此头足类动物中存在转录组多样性与基因组演化之间的权衡(Liscovitch-Brauer et al., 2017, Cell)。
另一方面,RNA编辑也可以弥补有害的G>A DNA突变。理论认为,一些位点基因组上发生G>A突变之后是轻微有害的,而RNA编辑则是由于容忍了这种有害DNA突变而得以保留下来(Jiang & Zhang, 2019, Nature Communications)。对灵长类RNA编辑位点的分析也支持了这一理论(An et al., 2019, Genome Biology)。总之,RNA编辑既可以用于增加多样性,又可以弥补有害突变,这两种功能的相对权重在不同的物种中并不相同,有待进一步研究。
最后,作者讨论了RNA编辑的趋同演化。趋同演化是指关系较远的物种独立演化出相似的特征以适应特定环境。在哺乳动物中,ADAR1介导的对重复序列的RNA编辑阻止MDA5引发免疫反应,该机制在哺乳动物中高度保守,但重复序列上的编辑位点在不同哺乳动物中保守性却很低。在果蝇和蜜蜂中,发生在CDS的编辑位点,都展现出适应性信号,这些位点虽然位置不保守,但很多发生在相同基因上(图四),因此RNA编辑在果蝇和蜜蜂两个进化枝上呈现出趋同演化(Duan et al., 2021, iScience)。

图四,果蝇和蜜蜂里RNA编辑的趋同演化(Duan et al., 2021, iScience)。
金沙威尼斯欢乐娱人城陆剑研究员为该论文的通讯作者。生科院博士后段元格是论文的第一作者,博士生唐小鹿也对该论文做出重要贡献。该项研究得到了科技部、国家自然科学基金委(NSFC)和以色列科学基金会(ISF)的支持。
原文链接:http://doi.org/10.1002/wrna.1666
参考文献
[1] Y. Duan, S. Dou, S. Luo, H. Zhang and J. Lu. Adaptation of A-to-I RNA editing in Drosophila. PLoS Genetics. 2017, 13: e1006648. https://doi.org/10.1371/journal.pgen.1006648
[2] Y. Duan, S. Dou, H. Zhang, C. Wu, M. Wu and J. Lu. Linkage of A-to-I RNA editing in metazoans and the impact on genome evolution. Molecular Biology and Evolution. 2018, 35: 132-148. https://doi.org/10.1093/molbev/msx274
[3] Y. Duan, S. Dou, H. Porath, J. Huang, E. Eisenberg, J. Lu. A-to-I RNA editing in honeybees shows signals of adaptation and convergent evolution. iScience. 2021, 24: 101983. https://doi.org/10.1016/j.isci.2020.101983
[4] N. Liscovitch-Brauer, S. Alon, H. Porath, B. Elstein, R. Unger, T. Ziv, A. Admon, et al. Trade-off between Transcriptome Plasticity and Genome Evolution in Cephalopods. Cell. 2017, 169: 191-202 e111. http://dx.doi.org/10.1016/j.cell.2017.03.025
[5] D. Jiang, & J. Zhang. The preponderance of nonsynonymous A-to-I RNA editing in coleoids is nonadaptive. Nature Communications, 2019, 10, 5411. https://doi.org/10.1038/s41467-019-13275-2
[6] N. An, W. Ding, X. Yang, J. Peng, B. He, Q. Shen, et al. Evolutionarily significant A-to-I RNA editing events originated through G-to-A mutations in primates. Genome Biology, 2019, 20, 24. https://doi.org/10.1186/s13059-019-1638-y